News
03.03.2021
Proteinsequenz-Clustering mit DIAMOND
In ihrer Bachelorarbeit erweiterte und optimierte Jasmin Katz den Clustering-Algorithmus von DIAMOND.
In den letzten Jahrzehnten kam es zu einem exponentiellen Anstieg an Proteinsequenzen aufgrund von Großprojekten, deren Ziel es war unbekannte Spezies zu sequenzieren. Die Anzahl an Proteinsequenzen wird auch in Zukunft weiter steigen, durch Großprojekte wie beispielsweise dem Earth BioGenome Project. Um diese große Menge an Daten effizient analysieren zu können, spielt Proteinsequenz-Clustering eine wichtige Rolle. Damit ist nicht nur die Reduktion großer Proteindatensätze möglich, sondern auch die Identifikation funktioneller und evolutionärer Gemeinsamkeiten verschiedener Proteine.
Der zum Clustern verwendete graphen-basierte Greedy-Vertex-Cover-Algorithmus wurde im Zuge der Bachelorarbeit um die Option des kaskadierten Clusterings erweitert. Zudem wurde der Ressourcenverbrauch durch externes Speichern des Knotengraphens begrenzt und individuell auf den Arbeitsspeicher des Nutzenden anpassbar gemacht.
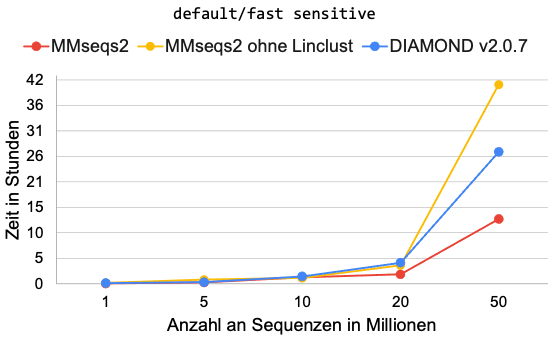
Mit den neu hinzugefügten Optionen wurde die Skalierbarkeit des Greedy-Vertex-Cover-Algorithmus auf zufälligen Stichproben der NR-Datenbank getestet. Die Ergebnisse von DIAMOND wurden mit dem Tool MMseqs2 verglichen, welches aktuell das schnellste und beste Tool zum Clustern großer Datensätze ist. Dabei konnte gezeigt werden, wenn die Parameter vergleichbar gehalten werden, dass DIAMOND schneller als MMseqs2 ist.