ZMBP News Archiv
02.12.2009
Gene und Proteine erklären nicht alles – neue Erkenntnisse verändern die Sicht der klassischen Genetik.
Andreas Wachter untersucht am Zentrum für Molekularbiologie der Pflanzen (ZMBP) der Universität Tübingen molekulare Regulationsmechanismen in zentralen biologischen Prozessen. Der Emmy Noether-Forschungsgruppenleiter überträgt neueste Erkenntnisse aus der Forschung an Bakterien erstmals auf Pflanzen
Gene und Proteine erklären nicht alles – neue Erkenntnisse verändern die Sicht der klassischen Genetik. [26.08.2009]
Andreas Wachter untersucht am Zentrum für Molekularbiologie der Pflanzen (ZMBP) der Universität Tübingen molekulare Regulationsmechanismen in zentralen biologischen Prozessen. Der Emmy Noether-Forschungsgruppenleiter überträgt neueste Erkenntnisse aus der Forschung an Bakterien erstmals auf Pflanzen
<link http: www.zmbp.uni-tuebingen.de zmbpinformation news pressdownloads emmynoether_wachter_final.pdf dr. andreas>Pressemitteilung als pdf-Datei
Eine Welt für sich
Andreas Wachter forscht in einer vermeintlich anderen Welt, der „RNA-Welt“. Und er ist überzeugt davon, dass die Entdeckungen aus dieser Welt elementare Bedeutung für das Verständnis der Steuerungs- und Regulationsprozesse in den Zellen aller Lebewesen hat: „Die Rolle der RNA für die Lebensprozesse in der Zelle wurde lange Zeit völlig unterschätzt! Es werden vermutlich sehr viel mehr Prozesse als wir bisher ahnen von der RNA selbst gesteuert.“
Viele Wissenschaftler betrachten die RNA nur als einen notwendigen Zwischenschritt, um die Erbinformation der DNA aus dem Zellkern zu übersetzen in Proteine, die in der Zelle alle lebenswichtigen Funktionen erfüllen. Viel mehr als eine solche Matrizenfunktion hat man der RNA lange nicht zugetraut, zumal sie mit ihren vier Bausteinen in einem Einzelstrang geradezu primitiv erscheint gegenüber den hochkomplexen und spezialisierten Strukturen, in denen Proteine in der Zelle vorkommen.
Tatsächlich aber übernimmt die RNA zahlreiche weitere Aufgaben in der Zelle und man deutet diese Fähigkeit als Überbleibsel aus einer sogenannten „RNA-Welt“, einer frühen Stufe der Evolution, in der die RNA sämtliche Funktionen von DNA und Proteinen mit übernahm. Wachter geht hier allerdings noch einen Schritt weiter: „In der Tat spricht viel für eine ursprüngliche RNA-Welt. Die ist jedoch keinesfalls verschwunden, sondern wurde lediglich durch DNA und Proteine ergänzt!“
Die dritte Dimension betrachten
Wachter hat für diese provokante These gute Argumente, die darauf beruhen, dass er die RNA nicht nur in ihrer eindimensionalen Abfolge der Basen auf einem Einzelstrang betrachtet, sondern auch deren Struktur in der zweiten und dritten Dimension untersucht. Und in dieser räumlichen Betrachtung entpuppt sich der vermeintliche Nachteil der RNA, die im Gegensatz zur Doppelhelix der DNA nur aus einem einzelnen Strang besteht, plötzlich als enormer Vorteil: Die Ausbildung von räumlichen Strukturen ist im Einzelstrang sehr viel einfacher. Und diese Faltungen der RNA vervielfachen die Möglichkeiten für Interaktionen mit anderen Molekülen – und mit sich selbst.
Erklärungen der klassischen Genetik unzureichend
Wachter untersucht diese besonderen Fähigkeiten der RNA an einem klassischen Mechanismus, dem sogenannten „Spleißen“, bei dem bestimmte Abschnitte aus den RNA-Molekülen ausgeschnitten und damit die Informationen auf dem Einzelstrang komprimiert werden. Dieser Vorgang findet hunderttausendfach pro Minute in jeder Zelle statt und ist für die Funktion der Lebensprozesse elementar. Bisher wurde angenommen, dass die komplexe Aufgabe der Erkennung und Entfernung von RNAAbschnitten nur von speziellen Proteinen geleistet werden kann. Wachter stellt diese klassische Interpretation in Frage: „Man ging bisher davon aus, dass die RNA nur eine passive Rolle spielt und alle Entscheidungen im Prozess des Spleißens von Proteinen getroffen werden. Eine sehr elegante Lösung wäre hingegen, wenn die RNA selbst diesen Prozess beeinflussen kann, ohne erst den Umweg über Proteine zu gehen.“ Auf der Suche nach einem solchen Mechanismus stieß Wachter auf die Untersuchungen zum „Alternativen Spleißen“ und erforschte diesen Prozess als Postdoc in der auf dem Gebiet sogenannter RNA-Schalter weltweit führenden Arbeitsgruppe von Ronald Breaker an der renommierten Yale University in Connecticut, USA.
Die RNA steuert sich selbst
Dabei stellte sich heraus, dass durch die Faltung der RNA in dreidimensionale Strukturen tatsächlich ein effizienter Regulationsmechanismus in vielen Auf- und Abbauprozessen der Zelle möglich wird. Die RNA muss nicht auf Proteine warten, sondern kann die Prozesse durch spezielle Faltungen selbst regulieren und über unterschiedliche Strukturen verschiedene Reaktionsabläufe in der Zelle selbst steuern. Damit erhält der Prozess des klassischen Spleißens einen alternativen Regulierungsmechanismus, mit dem die Zelle schnell und dynamisch auf veränderte Bedingungen reagieren kann. Dieses Alternative Spleißen erhöht die Geschwindigkeit und Plastizität der Reaktionen in der Zelle und ist darüber hinaus ein äußerst sparsamer Regulationsmechanismus, da hier nur das RNA-Molekül selbst involviert ist. „Wachter sagt für die Zukunft voraus: „Die bisherigen Arbeiten wiesen das enorme Potenzial von RNA in vielen zellulären Prozessen nach. Ich bin mir sicher, dass dieses Potenzial in allen Lebewesen sehr viel stärker genutzt wird, als bisher angenommen.“
Entwicklung von Antibiotika
Der Einfluss der RNA-Struktur auf die Steuerung von Prozessen in den Zellen wurde erstmals 2002 an Bakterien entdeckt, inzwischen wurden über 20 verschiedene Klassen solcher sogenannter RNA-Schalter beschrieben. Die Erforschung dieser Strukturen wird weltweit intensiv voran getrieben, da man neben dem grundlegenden Verständnis biologischer Prozesse auch das große Potential zur Entwicklung neuer Antibiotika erkannt hat: Ein Mechanismus, der so effizient die Regulation von lebenswichtigen Prozessen steuert, kann, so die Hoffnung der Forscher, gezielt verändert und gegen zunehmend resistente Bakterienstämme eingesetzt werden.
RNA-Schalter auch in Pflanzen
Wachter ist einer der ersten, der diese an Bakterien erforschten Mechanismen auf höhere Organismen überträgt und in Pflanzenzellen untersucht. Dabei konnte er bereits zwei neue Klassen von RNA-Schaltern beschreiben und in hochrangigen internationalen Wissenschaftszeitschriften publizieren. „Ich gehe davon aus, dass unsere Arbeiten erst den Anfang darstellen“, so Wachter, „denn wir vermuten, dass diese RNA-Schalter auch in pflanzlichen Zellen verbreitet sind und in vielen Prozessen eine wichtige Rolle in der Regulation übernehmen können.“
Die für eine Erforschung der komplexen Strukturen erforderlichen Techniken hat sich Wachter in Amerika angeeignet und kann diese nun in Tübingen einsetzen. Dabei setzt er auf die Zusammenarbeit mit anderen Arbeitsgruppen am Zentrum für Molekularbiologie der Pflanzen (ZMBP) und dem benachbarten Max-Planck-Institut für Entwicklungsbiologie.
Anreiz selbständige Forschergruppen
Diese Vernetzung der Forschung und die Zusammenarbeit über starre Fachgrenzen hinweg war ein wesentlicher Grund dafür, dass Wachter sich für das ZMBP in Tübingen als Standort für seine unabhängige Forschergruppe entschieden hat. Er findet hier, was er schon in Amerika als große Bereicherung für seine Forschung empfunden hat: „es gab sehr viel Austausch und den Freiraum für neue Ideen“. Ausdrücklich lobt Wachter in diesem Zusammenhang das Emmy Noether-Programm der Deutschen Forschungsgemeinschaft (DFG): „Die DFG hat damit ein international konkurrenzfähiges Instrument geschaffen, dass genau die richtigen Anreize und Freiheiten bietet und eine frühzeitige Selbständigkeit auf dem Weg zum Hochschullehrer ermöglicht.“
Bildmaterial
(Klick auf das Bild für bessere Auflösung)
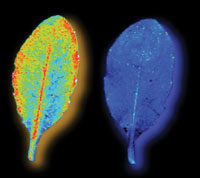
Einem RNA-Schalter bei der Arbeit zuschauen: Frische Blätter der Versuchspflanzen werden mit Laserlicht angeregt und anschließend mit hoher Auflösung in einem Fluoreszenzscanner aufgenommen. Unter normalen Bedingungen ist der Schalter an und die Blätter leuchten rot oder gelb. Nach Signalgabe wechselt der Schalter auf "Aus" und die Blätter erscheinen blau. Mit diesem Verfahren können die Wissenschaftler das Ein- und Ausschalten in lebenden Zellen und in Echtzeit beobachten.
<link http: www.zmbp.uni-tuebingen.de zmbpinformation news pressdownloads wachter_gruppe_jul09.jpg>
(Klick auf das Bild für bessere Auflösung)
Die Emmy Noether-Forschergruppe von Andreas Wachter (zweiter von links) am Zentrum für Molekularbiologie der Pflanzen.
<link http: www.zmbp.uni-tuebingen.de zmbpinformation news pressdownloads wachter_jul09.jpg andreas>
(Klick auf das Bild für bessere Auflösung)
Dr. Andreas Wachter, neuer Emmy Noether-Forschergruppenleiter am Zentrum für Molekularbiologie der Pflanzen.
Weiterführende Informationen
- <link http: www.zmbp.uni-tuebingen.de index.html for plant molecular>
- <link http: www.zmbp.uni-tuebingen.de zmbpinformation news pressnews>
www.zmbp.uni-tuebingen.de/ZMBPInformation/news/pressnews/090723emmy.html
- <link http: www.dfg.de forschungsfoerderung nachwuchsfoerderung emmy_noether>
www.dfg.de/forschungsfoerderung/nachwuchsfoerderung/emmy_noether/
Über Dr. Andreas Wachter
Andreas Wachter studierte Biologie mit einem Fokus auf Molekularbiologie, Botanik und Ökologie an der Universität Heidelberg. Nach einer Promotion auf dem Gebiet der molekularen Pflanzenwissenschaften in Heidelberg im Jahr 2004 folgten ein dreimonatiges Forschungsprojekt an der Harbin Universität in China sowie zwei Jahre als Postdoc an der Yale Universität, USA. Ab Sommer 2007 führte Wachter seine Forschung am Heidelberger Institut für Pflanzenwissenschaften fort und leitet seit 2009 eine Emmy Noether-Forschungsgruppe zum Thema „Alternatives Spleißen in Pflanzen“ am Zentrum für Molekularbiologie der Pflanzen an der Universität Tübingen. Alternatives Spleißen beschreibt einen wichtigen Schritt in der Reifung zellulärer Boten-RNAs und ist trotz seiner weiten Verbreitung in höheren Organismen bisher nur wenig erforscht.
Über das ZMBP
Das Zentrum für Molekularbiologie der Pflanzen (ZMBP) ist ein interfakultärer und interdisziplinärer Forschungsschwerpunkt an den Fakultäten für Biologie und für Chemie und Pharmazie der Universität Tübingen. Seit seiner Gründung 1999 nimmt das ZMBP in den Fachbereichen Genetik, Molekularbiologie, Biochemie, Zellbiologie, Physiologie und Entwicklungsbiologie von Pflanzen sowohl in der Forschung als auch in der Ausbildung von Nachwuchswissenschaftlern/ Innen eine internationale Spitzenstellung ein. Dies belegen eine Vielzahl von Publikationen in international führenden Wissenschaftsjournalen, ein hohes Drittmittelaufkommen, starkes Engagement in nationalen und internationalen Forschungsverbünden sowie die exzellenten Begutachtungen durch einen unabhängigen Wissenschaftlichen Beirat (ISAB). Mehr als 20 Rufe auf Professuren im In- und Ausland in den letzten acht Jahren sind ein deutlicher Beleg für das exzellente Forschungsumfeld – insbesondere für Nachwuchswissenschaftler.
Flache Hierarchien, Globalhaushalt, eigenverantwortliches Forschen in enger Verflechtung mit anderen Gruppen und die breite Unterstützung durch die Zentralen Service-Einrichtungen am ZMBP bieten optimale Rahmenbedingungen für Diplomanden, Doktoranden, Postdoktoranden und Gruppenleiter in insgesamt 16 unabhängigen Forschergruppen. Diese moderne Struktur, die ständige technologische Weiterentwicklung, und die Qualitätskontrolle durch den Wissenschaftlichen Beirat sichern die internationale Wettbewerbsfähigkeit und die wissenschaftliche Exzellenz des ZMBP.
Jüngste Anerkennung dieser Leistungen ist die Bewilligung eines neuen Forschungsgebäudes für das ZMBP durch den Wissenschaftsrat.
<link http: www.zmbp.uni-tuebingen.de>
und/oder Development & CommunicationMatthias Hendrichs (Tel.: 07071/29-73095, Email: mh@uni-tuebingen.de)
Über das Emmy-Noether-Programm der DFG
Mit dem Emmy Noether-Programm ermöglicht die Deutsche Forschungsgemeinschaft (DFG) jungen Forschern die frühe wissenschaftliche Selbständigkeit. Die Bewerber müssen über internationale Forschungserfahrung verfügen und herausragende Leistungen auf ihrem Arbeitsgebiet nachweisen.
Die DFG reagiert mit dem Emmy Noether-Programm auf den Abwanderungstrend deutscher Nachwuchswissenschaftler und bietet jungen Wissenschaftlern mit frühzeitiger wissenschaftlicher Unabhängigkeit und exzellenter Ausstattung einen attraktiven Anreiz zur Rückkehr nach Deutschland. Dabei haben die Wissenschaftler völlige Entscheidungsfreiheit bei der Auswahl der Hochschule, an der sie ihre Nachwuchsgruppe einrichten.
Benannt ist das Programm nach der deutschen Mathematikerin Amalie „Emmy“ Noether (1882 – 1935), Mitbegründerin der abstrakten Algebra und zugleich die erste Frau, die 1919 an der Universität Göttingen habilitierte. Sie wurde 1933, wie viele andere jüdische Wissenschaftler, von den Nationalsozialisten vertrieben und starb 1935 im amerikanischen Exil.
<link http: www.dfg.de forschungsfoerderung nachwuchsfoerderung emmy_noether>
www.dfg.de/forschungsfoerderung/nachwuchsfoerderung/emmy_noether/
Bitte senden Sie uns bei Veröffentlichung einen Beleg.
![]()
[Letzte Änderung: 28.08.2009]
Zurück