Aktuelles
26.01.2021
Gewebsspezifische Rekonstruktion restriktionsbasierter metabolischer Modelle basierend auf ReconX
In ihrer Masterarbeit implementierte Nantia Leonidou ein Tool zur gewebespezifischen Modellrekonstruktion und erstellte Wirt-Virus-Modelle, die für die COVID-19-Pandemie relevant sind.
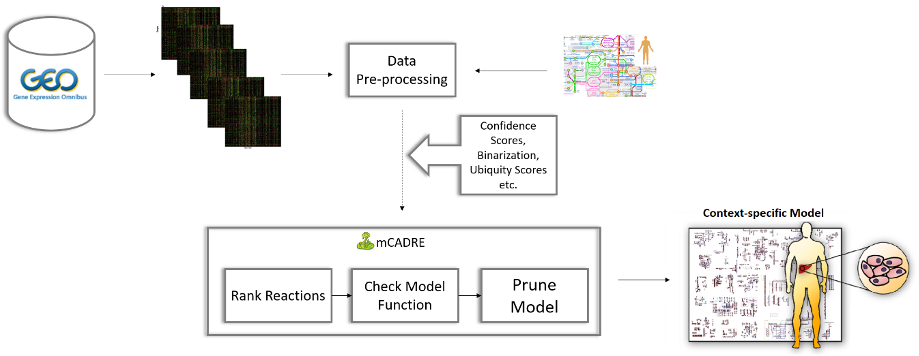
In einer Studie, die im Oktober 2007 veröffentlicht wurde, charakterisierten Wissenschaftler, die sich mit Coronaviren beschäftigen, die Situation in China als tickende „Zeitbombe” für einen möglichen Virusausbruch. Drei starke Indizien gaben ihnen Anlass zur Sorge: die tierbezogenen Essgewohnheiten in Südchina, das frühere Auftreten von SARS-CoV-ähnlichen Viren bei Hufeisennasen und die Fähigkeit von Coronaviren zur Rekombination. Achtzehn Jahre später erlebt die ganze Welt die Verwirklichung dieser Prophezeiung mit dem Auftreten von COVID-19, einer der tödlichsten Atemwegserkrankungen. In diesem Zusammenhang versuchen Wissenschaftler weltweit, die immunpathologische Reaktion des Wirts zu verstehen, wie sich das Virus an neue Wirte anpasst und wie es sich ausbreitet. Derzeit werden große Anstrengungen unternommen, um wirksame antivirale Behandlungen für die Coronaviren zu entdecken. Die Identifizierung potenzieller antiviraler Targets ist von großem Interesse und eine Möglichkeit, diese zu erkennen, ist die Analyse von Stoffwechselveränderungen in infizierten Zellen. Im Jahr 2012 veröffentlichten Wang et al. mCADRE (metabolic Context-specificity Assessed by Deterministic Reaction Evaluation), das darauf abzielt, gewebespezifische Modelle anhand von Genexpressionsdaten und Netzwerktopologie Informationen zu rekonstruieren. Der Algorithmus ist in MATLAB implementiert, und seine Funktionalität basiert ausschließlich auf der ersten Version des menschlichen Modells. Dies führte zu einer eingeschränkten Verwendbarkeit des Algorithmus, da neuere und umfassendere Modellversionen verfügbar sind.
In ihrer Masterarbeit erstellte Nantia Leonidou pymCADRE, eine Re-Implementierung von mCADRE in Python 3.8, und testete dessen Funktionalität mit allen drei derzeit verfügbaren Versionen des menschlichen Stoffwechselnetzwerks. Dabei wurde festgestellt, dass die mit fastFVA durchgeführten internen Optimierungen zu kontextspezifischen Modellen führten, die näher an der Grundwahrheit lagen.
Außerdem verbrauchen Viren Energie aus der Wirtszelle, um ihre eigene Massenproduktion zu steigern. Sie programmieren also Zellen genetisch um, um weitere Viruspartikel zu bilden und ihre eigene Vermehrung zu ermöglichen. Das Verständnis der metabolischen Basis der Wirt-Virus-Interaktion könnte genutzt werden, um die metabolischen Veränderungen und ihre Auswirkungen auf die Virusvermehrung vorherzusagen. Zu diesem Zweck wurden mit pymCADRE Wirts-Virus-Modelle (HVMs) erstellt, die helfen sollen, potentielle antivirale Targets gegen SARS-CoV-2 zu identifizieren. Mit diesen Modellen wurde die Existenz des kürzlich identifizierten potenziellen antiviralen Zielenzyms Guanylat-Kinase weiter verifiziert.