Aktuelles
23.05.2023
Rekonstruktion, Validierung und phänotypischen Vorhersagen stammspezifisches Stoffwechselmodells von Staphylococcus capitis.
Anna Lefarova erstellt in ihrer Bachelorarbeit ein metabolisches Modell für S. capitis.
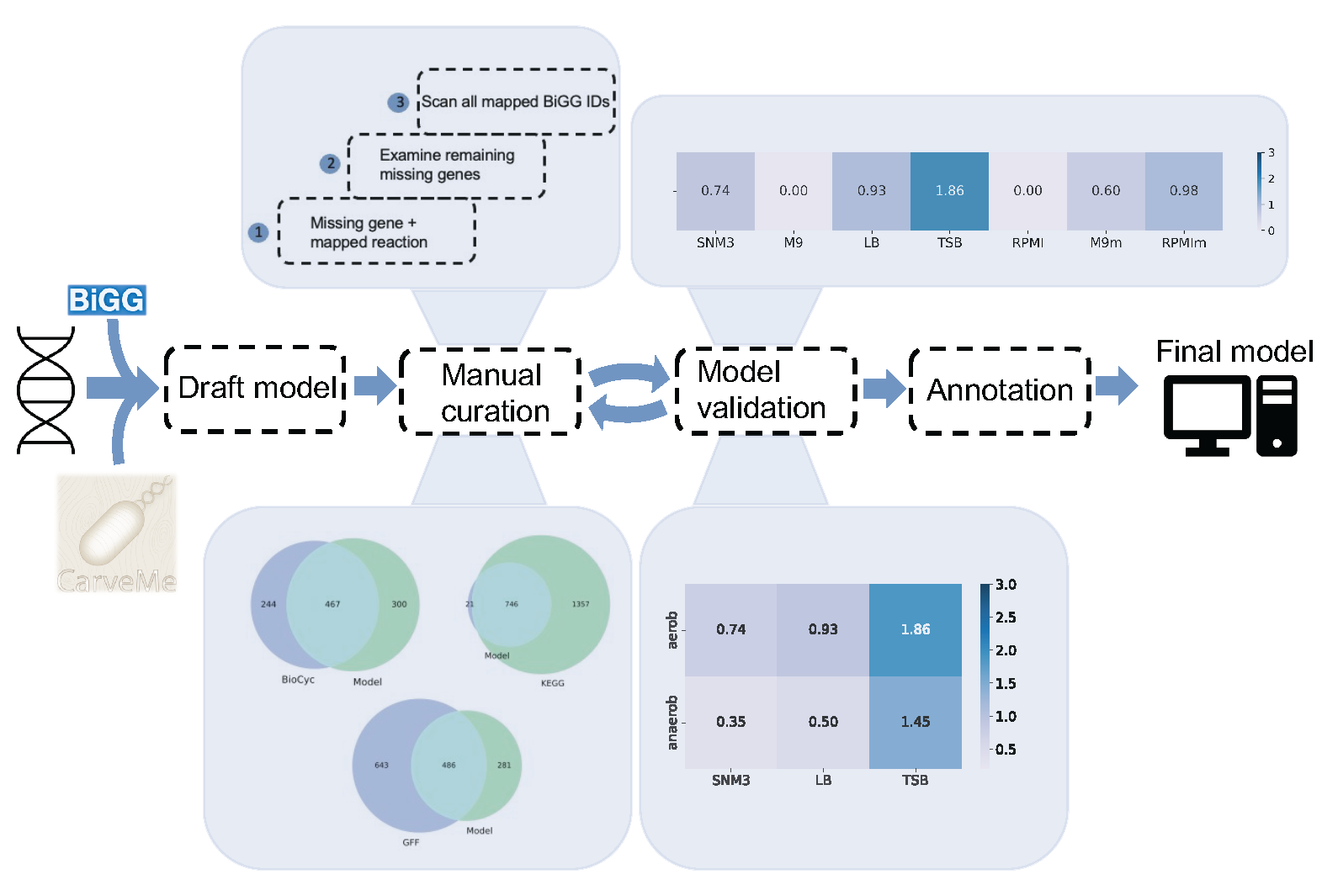
Staphylococcus capitis ist ein grampositives Bakterium, das zur Familie der koagulase-negativen Staphylokokken (CoNS) gehört. Einerseits wird es als vorteilhafte Hautmikrobiota angesehen, da es in der Lage ist, eine saure pH-Umgebung zu erzeugen und damit das Wachstum von pathogenen Mikroorganismen zu hemmen. Anderseits nimmt das klinische Interesse zum Bakterium zu, weil es als die Ursache für Spätinfektionen in Neugeborenen-Intensivstationen identifiziert wurde. Dabei wurde die Präsenz von S. capitis sowie an den medizinischen Oberflächen als auch in der Nasenhöhle des medizinischen Personals nachgewiesen. Das Bakterium verfügt über eine besondere Eigenschaft, die Biofilm-Bildung. Diese Fähigkeit ermöglicht das Überleben bei den ungünstigen Umweltbedingungen. Im Rahmen ihrer Bachelorarbeit „Analyse metabolischer Phänotypen von Staphylococcus capitis mittels restriktionsbasierter Modellierung” entwickelte Frau Anna Lefarova ein stammspezifisches Stoffwechselmodell auf Genomebene (GEM) des Organismus Staphylococcus capitis. Das Computer-Modell wurde nach einem hochstandardisierten Protokoll für die Rekonstruktionen im Genommaßstab erstellt und anhand verfügbaren Labordaten validiert. Darüber hinaus wurden die phänotypischen Vorhersagen bezüglich des Wachstums auf den unterschiedlichen Kohlenstoff- und Stickstoffquellen gemacht. Das erstellte Model wurde gegen automatisch generierte Modelle für S. capitis verglichen. Die manuell kuratierte metabolische Rekonstruktion stellt eine Grundlage für die in silico Simulationen und soll dazu beitragen, das Verständnis der metabolischen Fähigkeiten von S. capitis zu vertiefen.