Aktuelles
16.05.2023
Rekonstruktion, Vergleich und Wachstumssimulation stammspezifischer Stoffwechselmodelle von Staphylococcus haemolyticus
Gwendolyn O. Gusak entwickelt in ihrer Masterarbeit eine Sammlung hochwertiger Modelle für S. haemolyticus.
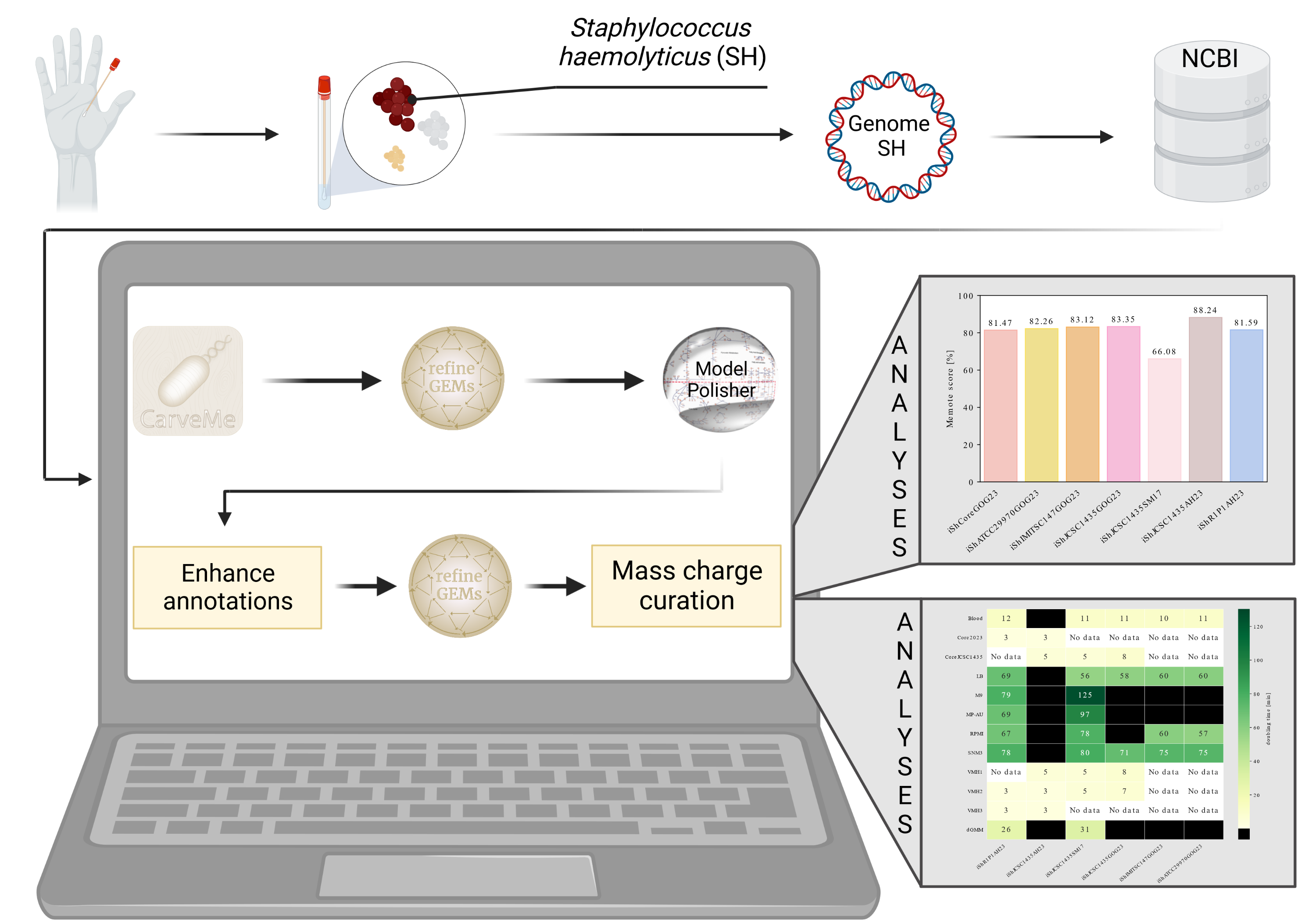
In Zeiten sich zunehmend ausbreitender Antibiotikaresistenzen gewinnt die umfassende Untersuchung opportunistischer Krankheitserreger zunehmend an Bedeutung. Sogenannte in-sillico-Ansätze (also im Computer durchgeführte biologische Experimente) wie die Simulation stammspezifisches Stoffwechselmodelle auf Genomebene (GEMs) leisten dazu einen wichtigen Beitrag, um Krankheitserregern zu untersuchen, da sie eine schnelle Prüfung plausibler Laborszenarien ermöglichen. Im Rahmen ihrer Masterarbeit „Rekonstruktion stammspezifischer Stoffwechselmodelle von Staphylococcus haemolyticus“ entwickelte Frau Gwendolyn O. Gusak drei neue GEMs des Organismus Staphylococcus haemolyticus (SH) und verglich diese mit für selbigen Organismus automatisch generierten GEMs. Ihr neuer Arbeitsablauf vom Genom zu einem GEM-Entwurf zeigt gute Ergebnisse, da 98,69% aller Proteinsequenzen gültige Identifikatoren der NCBI-Datenbank erhalten haben. Ihr resultierendes Modell iShIMITSC147GOG23 wies im Vergleich zu den anderen Modellen von SH ähnliche Ergebnisse auf. Darüber hinaus erweiterte sie im Rahmen ihrer Arbeit die Skript-Sammlung namens refineGEMs um einen Algorithmus, der Lücken in den Modellen füllt, woran sie auch nach Abschluss ihrer Arbeit weiter arbeiten möchte. Der Vergleich der MEMOTE-Punktzahlen der neuen Modelle mit den automatisch generierten Modellen zeigt, dass die Qualität der Modelle gleich hoch ist, mit Ausnahme des ältesten Modells aus dem Jahr 2017, das eine niedrige MEMOTE-Punktzahl von 66,08 % aufweist. Die Ergebnisse ihrer Wachstumssimulationen zeigen meist Verdopplungszeiten in einem für die Laborforschung plausiblen Bereich. Ihre Arbeiten an der Software als auch die Modelle selbst stellt sie öffentlich zugänglich auf GitHub sowie in der für die Systembiologie üblichen BioModels-Datenbank bereit. Nach Abschluss dieser Arbeit plant Frau Gusak, ihre Ergebnisse durch eine Fachzeitschrift begutachten und veröffentlichen zu lassen.