Aktuelles
17.05.2023
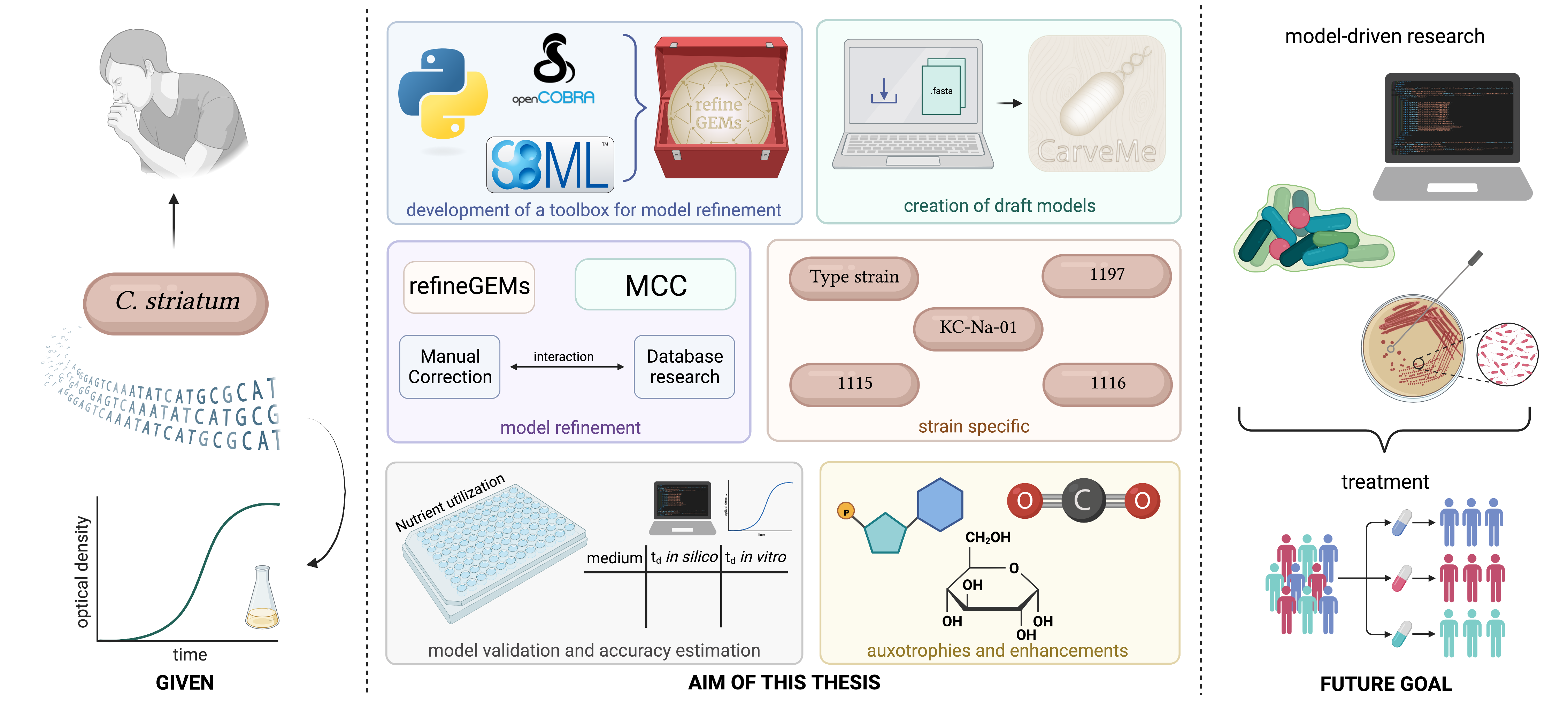
Stammspezifische Stoffwechselmodelle von Corynebacterium striatum
Famke Bäuerle rekonstruiert Stoffwechselmodelle und validiert diese in vitro im Rahmen zweier Masterarbeiten.
Antibiotikaresistente Bakterien führen zu immer mehr Infektionen weltweit. Eine Infektion mit dem Bakterium Corynebacterium striatum wurde, laut einer ungarischen Studie, seit der COVID-19-Pandemie sogar noch häufiger mit der Notwendigkeit der Beatmung von Patienten in Zusammenhang gebracht. Dennoch ist es weitestgehend unerforscht, und die Mechanismen, die es vom Bestandteil des normalen Mikrobioms des Menschen zum Krankheitserreger werden lassen, unbekannt.
Um mehr Einblicke in das Stoffwechselverhalten und mögliche Pathogenitätsfaktoren zu erhalten, können Stoffwechselmodelle verwendet werden. In einem Projekt für zwei Masterarbeiten hat Famke Bäuerle fünf stammspezifische genomskalige Stoffwechselmodelle von C. striatum erstellt und mittels Laborexperimenten validiert. Mittels zweier experimenteller Methoden wurde festgestellt, dass die Modelle auf vier verschiedenen Medien (LB, RPMI, M9 und CGXII) unterschiedlich gute Vorhersagen bieten.
Parallel dazu wurde das Python-Paket refineGEMs (github.com/draeger-lab/refinegems) entwickelt, welches andere Forscher zum Untersuchen und Kuratieren von Stoffwechselmodellen verwenden können.
Das Python-Paket und die Modelle wurden im Rahmen eines Vorabdrucks bereits auf BioRxiv veröffentlicht.