attempto online Forschung
02.06.2021
SARS-CoV-2: Ein Computermodell macht mögliche Angriffspunkte von Virus und Virusmutanten sichtbar
Wirksame Medikamente gegen das neue Coronavirus werden immer noch dringend benötigt. Dies umso mehr, als das Aufkommen von Virusmutanten die Impfstoffe an ihre Grenzen bringen könnte. Andreas Dräger, Forschungsgruppenleiter im Exzellenzcluster „Kontrolle von Mikroorganismen zur Bekämpfung von Infektionen“ (CMFI) der Universität Tübingen und DZIF-Wissenschaftler, arbeitet seit mehr als einem Jahr an einem Computermodell, das Schwachstellen und damit mögliche Angriffspunkte des Virus erkennt. So identifizierte er bereits ein menschliches Enzym als möglichen Angriffspunkt. In seiner neuesten Studie bestätigt sich dieses Ergebnis und der Bioinformatiker macht neue Angriffspunkte sichtbar und zeigt, dass sie auch die momentan verbreiteten Mutanten betreffen.
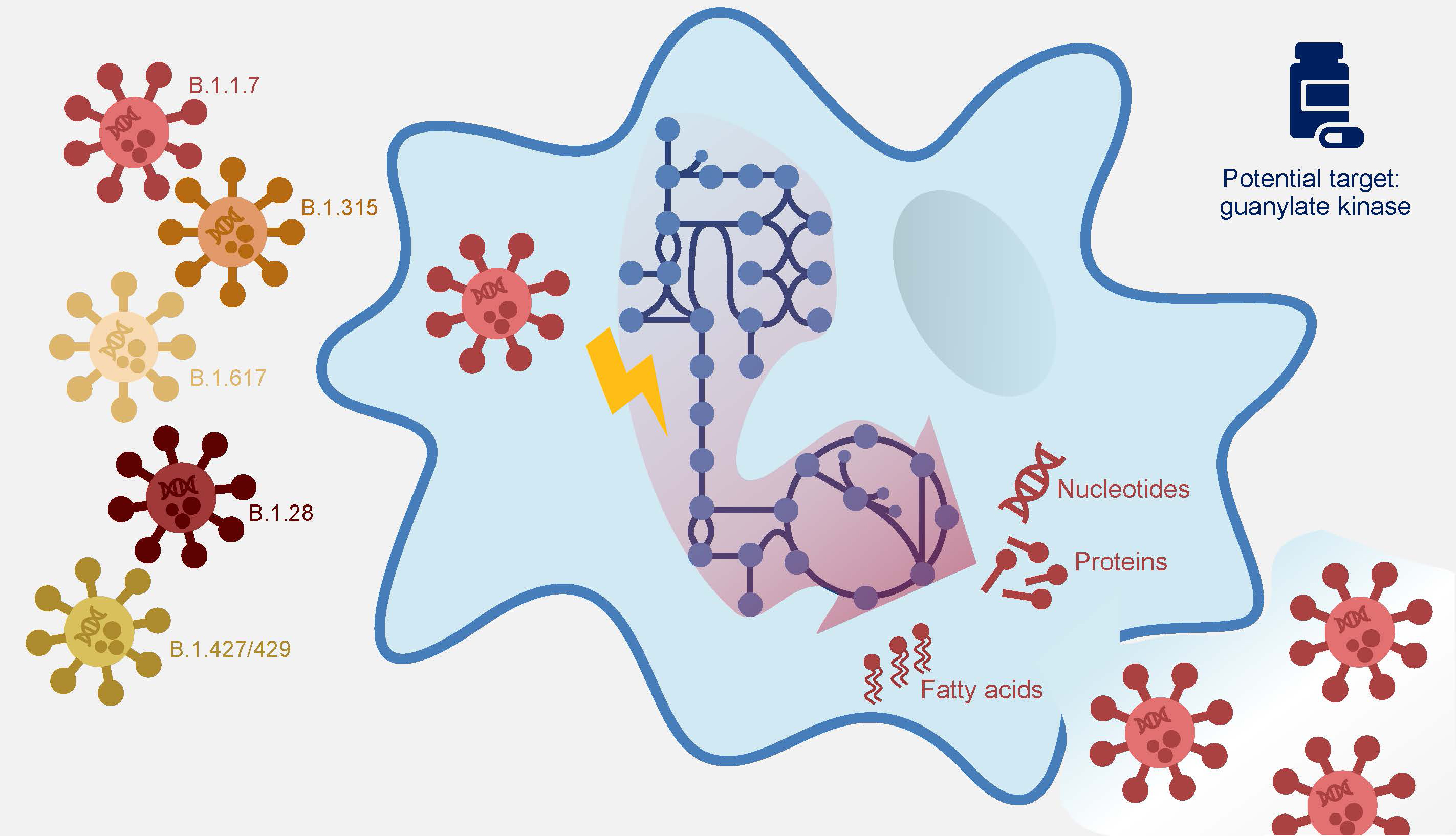
„Wir haben schon Anfang des Jahres in unserem Modell gesehen, dass ein menschliches Enzym – die Guanylatkinase 1 – für die Virusvermehrung unabdingbar ist“, erzählt Dr. Andreas Dräger. Werde diese Kinase ausgeschaltet, könne das Virus sich nicht mehr vermehren. „Die Zelle aber wird nicht geschädigt, was für einen möglichen Wirkstoff von großer Bedeutung ist“, ergänzt Dräger. Mit einer Juniorprofessur des DZIF an der Uni Tübingen betreibt der Bioinformatiker rechnerbasierte Systembiologie. Für diesen Ansatz entwickelte er gemeinsam mit seinen Teamkolleginnen Alina Renz und Lina Widerspick ein integriertes Computermodell weiter, das mit dem neuartigen Coronavirus SARS-CoV-2 und menschlichen Zellen, den Alveolarmakrophagen arbeitet. Letztere sind in den Lungenbläschen für die Abwehr von Fremdstoffen zuständig.
Ein Stoffwechselmodell auf Genomebene
Die Ausgangssituation im Modell ist, dass das Virus in den Wirt, hier die menschliche Alveolarmakrophage, eingedrungen ist und diese bereits umprogrammiert hat. Das Modell geht nun davon aus, dass das Virus neue Viruspartikel herstellen und sich ausbreiten will. Dazu nutzt es Materialien aus dem Wirt und zwingt die Wirtszellen zur Produktion neuer Virusbestandteile. „Wenn man die Zusammensetzung des Virus kennt, kann man verschiedene Szenarien durchspielen und sehen, wie sich die biochemischen Reaktionen in den Wirtszellen während einer Virusinfektion verändern.“
Mehr Wissen über das Virus verbessert das Modell
In der aktuellen Studie konnte die Forschungsgruppe ihr Computermodell mit neuen Informationen zu den Strukturproteinen und zum Fettstoffwechsel von SARS-CoV-2 verfeinern und damit auch neue mögliche Angriffsziele im Nukleotid- und Lipidstoffwechsel identifizieren. Darüber hinaus ergänzte er die Informationen zu den neuen Virusmutanten und analysierte, ob auch hier die bereits erkannten Schwachpunkte vorhanden waren. Das Ergebnis war bei allen getesteten Mutanten gleich: Wurde die Gyanylatkinase 1 (GK1) ausgeschaltet, wurde die Vermehrung des Virus gestoppt.
Das Team um Andreas Dräger geht davon aus, dass diese Ergebnisse eine wichtige Grundlage für die Entwicklung von Hemmstoffen des neuen Coronavirus darstellen. Sowohl die Guanylatkinase 1 als auch einige der gefundenen Wirkstoffziele im Nukleotid- oder Fettstoffwechsel könnten auf antivirale Substanzen ansprechen, ohne dem Menschen zu schaden. „Es sind bereits einige Hemmstoffe des Enzyms bekannt und wir wollen nun möglichst bald mit unserem Hamburger Kooperationspartner Dr. Bernhard Ellinger vom Fraunhofer IME ScreeningPort (IME) bereits zugelassene Hemmstoffe auf ihre Wirksamkeit gegen das neue Coronavirus testen“, erklärt Andreas Dräger.
Pressemitteilung Deutsches Zentrum für Infektionsforschung (DZIF)
Publikation:
Renz A, Widerspick L, Dräger A. Genome-Scale Metabolic Model of Infection with SARS-CoV-2 Mutants Confirms Guanylate Kinase as Robust Potential Antiviral Target. Genes 12: 796. (2021) doi: https://doi.org/10.3390/genes12060796.
Kontakt:
Dr. Andreas Dräger
Universität Tübingen
Exzellenzcluster „Kontrolle von Mikroorganismen zur Bekämpfung von Infektionen“ (CMFI)
Institut für Biomedizinische Informatik (IBMI) Rechnerbasierte Systembiologie der Infektionen und antimikrobiell-resistenten Krankheitserreger
+49 70 71 29-70459
draegerspam prevention@informatik.uni-tuebingen.de