attempto online
12.11.2020
Neue Veröffentlichung in Nature: Maschinelles Lernen hilft bei der Entschlüsselung des Gehirns
Ein internationales Team von Wissenschaftlern um Philipp Berens, Sprecher des Exzellenz-Clusters Maschinelles Lernen: Neue Perspektiven für die Wissenschaft der Universität Tübingen, und Andreas Tolias vom Baylor College in Houston, USA, hat einen neuen Artikel in Nature veröffentlicht. Darin befassen die Wissenschaftler sich mit der Physiologie und der Anatomie genetisch definierter Zelltypen im Gehirn. Mit Verfahren des Maschinellen Lernens haben sie multimodale Datensätze zusammengeführt und so festgestellt, dass genetisch definierte Zelltypen grob betrachtet zwar „Familien“ bilden, dass es aber innerhalb dieser Familien eine überraschend große Vielfalt an physiologischen und anatomischen Eigenschaften der Zellen gibt.
Die Veröffentlichung ist Teil der Arbeit des sogenannten BRAIN-Initiative Cell Census Network (BICCN) , das von den National Institutes of Health (NIH), einer Behörde des US-amerikanischen Gesundheitsministeriums, finanziert wird. Ziel der Initiative ist es, die zelluläre Taxonomie von Neuronen im Kortex von Mäusen, Affen und Menschen vollständig zu charakterisieren. Sie erwarten, dadurch bessere Ansatzpunkte für die Entwicklung von Therapien für neuropsychiatrische und neurodegenerative Erkrankungen zu liefern.
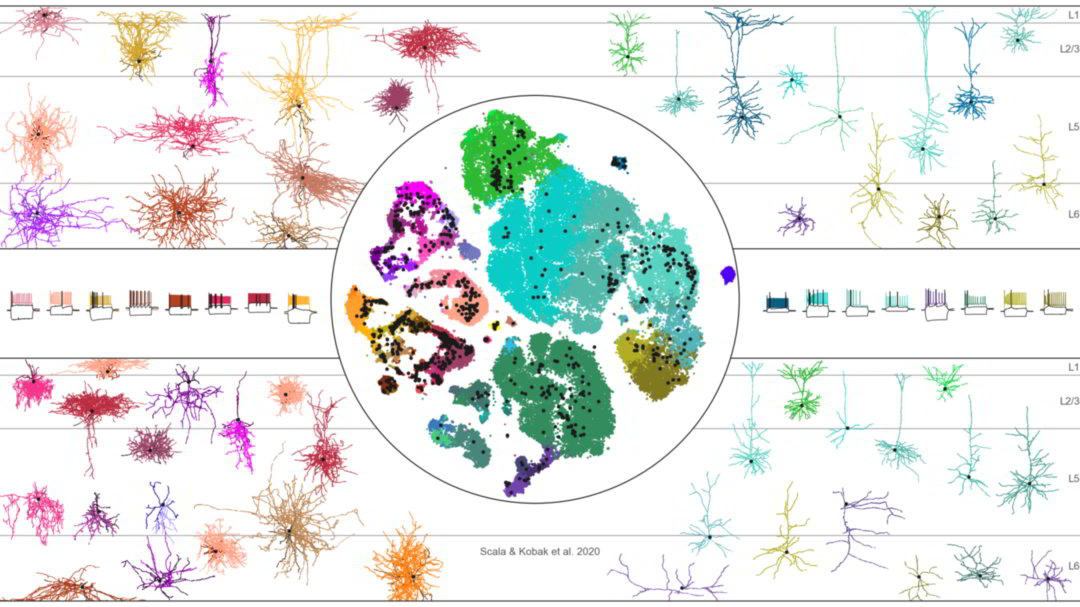
Tiere können in Arten gruppiert werden und diese über ihre Abstammung und ihre Verwandtschaftsbeziehungen zueinander zum "Baum des Lebens" zusammengefügt werden. Analog dazu nimmt man an, dass Neuronen im Gehirn verschiedene Zelltypen bilden, die wiederum in eine Hierarchie neuronaler Familien und Klassen eingeordnet werden können. Nach der derzeitigen Auffassung zeichnet sich ein neuronaler Zelltyp durch ein gemeinsames genetisches Profil aus, aus dem sich eindeutige physiologische und anatomischen Eigenschaften und auch Verbindungsmuster ergeben. Mit Hochdurchsatz-Einzelzell-Sequenziertechniken konnten zuletzt Dutzende neuronaler Typen auf der Grundlage ihrer genetischen Profile identifiziert werden. Es war allerdings schwierig, die notwendigen Daten dafür zu sammeln, um auch die Anatomie und Physiologie dieser genetischen Typen zu beschreiben. Das ist jedoch entscheidend, wenn man ihre Rolle im gesunden Gehirn und bei seinen Erkrankungen verstehen möchte.
Für ihre Studie haben die Wissenschaftler die technisch herausfordernde, sogenannte Patch-Seq-Technik verwendet, um eine große Datenbank mit multimodalen Daten aufzubauen. Die Datenbank enthält genetische, anatomische und physiologische Informationen von mehr als 1.300 Zellen aus dem motorischen Kortex der Maus. Mithilfe von Techniken des maschinellen Lernens stellten die Forscher fest, dass breite genetische "Familien" von Neuronen auch weitgehend unterschiedliche Phänotypen aufwiesen, dass aber innerhalb dieser Gruppen die anatomischen und physiologischen Eigenschaften kontinuierlich variierten. Hervorzuheben ist, dass einige genetische Typen auch stark variierende Eigenschaften aufwiesen. In einer vergleichbaren Studie, die gleichzeitig in Cell veröffentlicht wurde, kamen Wissenschaftler des Allen-Instituts in Seattle, USA, zu ähnlichen Ergebnissen.
Dmitry Kobak von der Universität Tübingen, leitender Data Scientist der neuen Veröffentlichung in Nature, erklärt in Anspielung auf den „Baum des Lebens“: „Wir denken, dass der 'Baum der kortikalen Zelltypen' eher wie ein Bananenbaum mit wenigen großen Blättern aussehen könnte als wie ein Olivenbaum mit vielen kleinen Blättern". In dieser Metapher bleibend, folgen die Neuronen also einer Hierarchie, die auf der Ebene der Familien (den großen Blättern) aus verschiedenen, sich nicht überlappenden Zweigen besteht. Innerhalb jedes Bananenblattes existiert jedoch eine Bandbreite an Zellen, die fortlaufende und miteinander korrelierende genetische, anatomische und physiologische Variationen aufweisen.
Originalveröffentlichung: https://www.nature.com/articles/s41586-020-2907-3
Philipp Berens und Theresa Authaler
Kontakt:
Prof. Dr. Philipp Berens
Eberhard Karls Universität Tübingen
Forschungsinstitut für Augenheilkunde
Werner Reichardt Centrum für Integrative Neurowissenschaften (CIN)
E-Mail: philipp.berensspam prevention@uni-tuebingen.de
Pressekontakt:
Theresa Authaler
Eberhard Karls Universität Tübingen
Exzellenzcluster „Maschinelles Lernen: Neue Perspektiven für die Wissenschaft“
Referentin für Medien- und Öffentlichkeitsarbeit
E-Mail: theresa.authalerspam prevention@uni-tuebingen.de